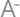 |
|
 |
|

图说:外切酶Rat1与Pol II形成的复合物结构 采访对象供图(下同)
新民晚报讯(记者 郜阳)遗传中心法则描述遗传信息在DNA中储存,经过RNA聚合酶传递到中间介质mRNA,随后被核糖体解码翻译成蛋白质。RNA聚合酶以DNA为模板合成RNA的过程称为基因转录,它是基因表达的第一步,也是基因表达调控的重要环节。
基因转录合成mRNA的过程分为起始,延伸和终止三个阶段。近年来mRNA起始合成和延伸的机制逐渐被揭开。然而,由于转录终止的动态和不稳定特性,其机制仍不清楚。转录终止是停止mRNA延伸、释放mRNA、并从DNA上解离的过程。正确的转录终止对mRNA的稳定性和RNA聚合酶的高效循环利用至关重要。
北京时间今天凌晨,中国科学院分子植物科学卓越创新中心/中国科学院合成生物学重点实验室张余研究组在国际顶尖学术期刊《自然》(Nature)上发表最新研究成果,揭示了酵母细胞mRNA转录终止的分子机制。值得一提的是,就在本月1日,张余研究团队还在国际顶尖期刊《细胞》(Cell)封面发表研究成果。
据介绍,真核细胞RNA聚合酶II(Pol II)的 mRNA转录终止机制非常保守,其依赖一个RNA外切酶的活性。针对真核细胞mRNA转录终止的机制已经被研究了多年,提出了包括“鱼雷”“异构”等多个模型,但是其机制尚存争议——异构模型认为,核酸外切酶结合诱导Pol II构象变化,促进mRNA自发解离;鱼雷模型认为,核酸外切酶切割下游RNA并追赶Pol II,赶上后触发mRNA解离。转录终止具体通过何种机制尚存争论。
研究团队在体外重构了外切酶终止Pol II mRNA合成的过程、并解析了外切酶结合Pol II的转录复合物三维结构。科研人员发现外切酶稳定结合在Pol II的mRNA通道外侧,直接将Pol II合成的RNA引导到外切酶的活性中心,并且外切酶的结合能够促进Pol II转录延伸因子的解离,延缓Pol II的转录延伸速率。据此,研究团队提出了外切酶介导真核细胞mRNA转录终止的分子机制。
张余介绍,该项研究成功解析了真核生物mRNA转录终止状态的复合物结构,揭示了外切酶终止Pol II mRNA合成的分子机制,对理解基因转录的工作机制具有重要意义。

图说:论文通讯作者张余(左)与第一作者曾媛
中国科学院分子植物科学卓越创新中心张余组博士生曾媛(已毕业)为论文的第一作者,张余研究员为通讯作者。本课题受到上海市基础研究特区计划,科技部重点研究计划资助。
新民报系成员|客户端|官方微博|微信矩阵|新民网|广告刊例|战略合作伙伴
北大方正|上海音乐厅|中卫普信|今日头条|钱报网|中国网信网|中国禁毒网
增值电信业务经营许可证(ICP):沪B2-20110022号|互联网新闻信息服务许可证:31120170003|信息网络传播视听节目许可证:0909381
广电节目制作经营许可证:(沪)字第536号|违法与不良信息举报电话15900430043|跟帖评论自律管理承诺书
 |沪公网安备 31010602000044号|沪公网安备 31010602000590号|沪公网安备 31010602000579号
|沪公网安备 31010602000044号|沪公网安备 31010602000590号|沪公网安备 31010602000579号
新民晚报官方网站 xinmin.cn ©2024 All rights reserved